はじめに #
Pythonの機械学習用ライブラリScikit-learnに実装されている、スケール変換について調べた。スケール変換を行うクラス3つのパラメータとメソッドをまとめ、各変換の結果を比較した。
スケール変換は、扱う数値データを何らかの規則で変換するものである。機械学習で桁数の異なるデータをまとめて扱うときには、スケール変換がほぼ必須となる。 通常、ニューラルネットワークやSVM(サポートベクターマシン)では、スケール変換をしないとなかなか学習が進まない。ただし、ランダムフォレスト等の決定木を使う手法ではスケール変換は不要である。
この記事では、以下3つのスケール変換方法を扱う。
- StandardScaler: 標準化(平均0, 分散1)
- RobustScaler: 外れ値に頑健な標準化
- MinMaxScaler: 正規化(最大1, 最小0)
2021/01/28 標準偏差と分散を誤って記述している箇所があったため修正。
環境 #
記事執筆時点で使用したライブラリのバージョンは以下の通り。
| ソフトウェア | バージョン |
|---|---|
| Python | 3.6.5 |
| Scikit-learn | 0.19.1 |
| NumPy | 1.14.3 |
| matplotlib | 2.2.2 |
Pythonで以下の通りライブラリをインポートする。Scikit-learnのpreprocessingモジュールにスケール変換処理がまとめられている。
import numpy as np
import matplotlib.pyplot as plt
from sklearn import preprocessing
以下、各スケール変換のパラメータとメソッドについてまとめた結果を記す。
標準化(平均0, 分散1)する #
データの平均値と分散を変換する操作を標準化と呼ぶ。平均値を0, 分散を1とすることが多い。変換操作は以下の式で表される。
$$ Y = \frac{X-\mu}{\sigma} $$ここで、\(Y\)は変換後のデータ、\(X\)は変換前のデータである。また、\(\mu, \sigma\)は、それぞれ\(X\)の平均、標準偏差である(\(\sigma^2\)は分散)。
Scikit-learnで標準化は、
関数としてはscale,
クラスとしてはStandardScaler
という名前で用意されている。
StandardScalerクラスを使うと、あるデータに対して行った変換を別のデータに対して適用できる。
機械学習の場合、学習データに対して行った変換を、検証データに対して行うので、実用上はStandardScalerを使う機会が多いと思う。そのため、本記事ではStandardScalerのみ扱う。
StandardScalerのパラメータ #
preprocessing.StandardScaler(copy=True, with_mean=True, with_std=True)
StandardScalerクラスの主なパラメータの説明は以下の通り。基本的に全てデフォルトのまま使う。
copy
ブール型。デフォルト値はTrue.
Falseの場合、transformやfit_transformメソッドで変換時に、変換元のデータを破壊的に変換する。Trueの場合、元のデータは変換されない。
with_mean
ブール型。デフォルト値はTrue.
Trueの場合、平均値を0とする。
Falseの場合、以下の変換になる。
\(Y = \frac{X}{\sigma}\)
分散は1になるが、平均が維持されるとは限らない。
with_std
ブール型。デフォルト値はTrue.
Trueの場合、分散を0とする。
Falseの場合、以下の変換になる。
\(Y = X-\mu\)
分散は変化せず、平均は0となる。
StandardScalerのメソッド #
良く使うメソッドは次の3つ。
fit(X)
配列Xの平均と標準偏差を計算して、記憶する(変換は行わない)。
transform(X)
配列Xに変換を施して、変換後の配列を返す。
fit_transform(X)
配列Xに対して、fitとtransformを同時に行う。
なお、2次元配列を変換する場合、縦 (axis=0) 方向に変換が行われる。
StandardScalerの使用例 #
以下の2次元配列xを用意する。
x = np.arange(0, 8, 1.).reshape(-1, 2)
print(x)
print(x.mean(axis=0))
print(x.std(axis=0))
実行結果:
[[0. 1.]
[2. 3.]
[4. 5.]
[6. 7.]]
[3. 4.]
[2.23606798 2.23606798]
第0列目の平均は3, 標準偏差は2.24, 第1列目の平均は4, 標準偏差は2.24である。
次に、xを標準化する。
sscaler = preprocessing.StandardScaler() # インスタンスの作成
sscaler.fit(x) # xの平均と標準偏差を計算
y = sscaler.transform(x) # xを変換
print(y)
print(y.mean(axis=0))
print(y.std(axis=0))
実行結果
以下の通り、変換後の配列yは各列とも平均は0, 標準偏差は1となった。
[[-1.34164079 -1.34164079]
[-0.4472136 -0.4472136 ]
[ 0.4472136 0.4472136 ]
[ 1.34164079 1.34164079]]
[0. 0.]
[1. 1.]
ここで、
sscaler.fit(x) # xの平均と標準偏差を計算
y = sscaler.transform(x) # xを変換
は、以下と同じである。
y = sscaler.fit_transform(x) # xを変換した結果が返る
外れ値に頑健な標準化 #
変換前のデータに極端に大きな値または小さな値が含まれていた場合、標準化を行うと大きく結果が変わってしまう。これを避けるため、データの四分位点を基準にして標準化を行う方法がある。
Scikit-learnでこのような変換は、
関数としてはrobust_scale,
クラスとしてはRobustScaler
という名前で用意されている。
本記事ではRobustScalerのみ扱う。
RobustScalerの変換操作は以下の式で表される。
ここで、\(Y\)は変換後のデータ、\(X\)は変換前のデータである。 また、\(Q_1, Q_2, Q_3\)は、それぞれ\(X\)の第1~第3四分位点である。 標準化と比較すると、元のデータの平均が\(Q_2\)(中央値)、標準偏差が\(Q_3-Q_1\)であると仮定しているとも考えられる。 なお、上の式の分母で、どの範囲(パーセンタイル)のデータを使うかは設定で変更可能である。
RobustScalerのパラメータ #
preprocessing.RobustScaler(with_centering=True, with_scaling=True,
quantile_range=(25.0, 75.0), copy=True)
RobustScalerクラスの主なパラメータの説明は以下の通り。
外れ値の多さに対して、quantile_rangeを変更する。
with_centering
ブール型。デフォルト値はTrue.
Trueの場合、データから中央値を引いて、平均を0とする。
with_std
ブール型。デフォルト値はTrue.
Trueの場合、quantile_rangeで選択したパーセンタイルのデータの差でデータを割る。
quantile_range
タプル型。デフォルト値は(25.0, 75.0).
標準化を行うデータの範囲をパーセンテージで指定する。
(25.0, 75.0)の場合、下位25%と上位25%にある値の差でデータ全体を割る。
また、特徴量が複数ある場合、それぞれの特徴量に対して数値が選ばれる。
なお、データの値は、NumPyのpercentile関数で取得している。
この関数は、パーセンタイルとデータ数が一致しない場合、補間して返す。そのため、データ数が少ない場合や、離散的な場合は注意する。
copy
ブール型。デフォルト値はTrue.
Falseの場合、transformやfit_transformメソッドで変換時に、変換元のデータを破壊的に変換する。
Trueの場合、元のデータは変換されない。
RobustScalerのメソッド #
StandardScalerと同じく、良く使うメソッドは次の3つ。
fit(X)
transform(X)
fit_transform(X)
RobustScalerの使用例 #
以下の2次元配列xを用意する。
x = np.arange(0, 8, 1.).reshape(-1, 2)
print(x)
print(x.mean(axis=0))
print(x.std(axis=0))
実行結果:
[[0. 1.]
[2. 3.]
[4. 5.]
[6. 7.]]
[3. 4.]
[2.23606798 2.23606798]
- 第0列目の平均は3, 標準偏差は2.24,
- 第1列目の平均は4, 標準偏差は2.24
である。
次に、xを標準化する。下位25%と上位25%、すなわち[0. 1.]と[6. 7.]が無視される。
rscaler = preprocessing.RobustScaler(quantile_range=(25., 75.))
rscaler.fit(x)
y = rscaler.transform(x[1:3])
print(y)
print(y.mean(axis=0))
print(y.std(axis=0))
実行結果:以下の通り、変換後の配列yは各列とも平均は0, 標準偏差は0.745となった。
[[-1. -1. ]
[-0.33333333 -0.33333333]
[ 0.33333333 0.33333333]
[ 1. 1. ]]
[0. 0.]
[0.74535599 0.74535599]
正規化(最大1, 最小0)する #
データの最大値と最小値を制限する変換を正規化と呼ぶ。最大値を1, 最小値を0とすることが多い。 変換操作は以下の式で表される。
$$Y = \frac{X-x_{\min}}{x_{\max}-x_{\min}}$$ここで、\(Y\)は変換後のデータ、\(X\)は変換前のデータである。 また、\(x_{\min}, x_{\max}\)は、それぞれ\(X\)の最小値、最大値である。
Scikit-learnで正規化は、
関数としてはminmax_scale,
クラスとしてはMinMaxScaler
という名前で用意されている。
標準化と同様に、本記事ではMinMaxScalerクラスのみ扱う。
MinMaxScalerのパラメータ #
preprocessing.MinMaxScaler(feature_range=(0, 1), copy=True)
MinMaxScalerクラスの主なパラメータの説明は以下の通り。
feature_range
タプル型。デフォルト値は(0, 1).
変換後の最大値、最小値を設定する。
copy
ブール型。デフォルト値はTrue.
Falseの場合、transformやfit_transformメソッドで変換時に、
変換元のデータを破壊的に変換する。
Trueの場合、元のデータは変換されない。
MinMaxScalerのメソッド #
StandardScalerと同じく、良く使うメソッドは次の3つ。
fit(X)
transform(X)
fit_transform(X)
MinMaxScalerの使用例 #
標準化と同じ2次元配列xを用意する。
x = np.arange(0, 6, 1.).reshape(-1, 2)
print(x)
print(x.min(axis=0))
print(x.max(axis=0))
実行結果:
[[0. 1.]
[2. 3.]
[4. 5.]]
[0. 1.]
[4. 5.]
第0列目の最小値は0, 最大値は4, 第1列目の最小値は1, 最大値は5である。
次に、xを正規化する。
mmscaler = preprocessing.MinMaxScaler() # インスタンスの作成
mmscaler.fit(x) # xの最大・最小を計算
y = mmscaler.transform(x) # xを変換
print(y)
print(y.min(axis=0))
print(y.max(axis=0))
実行結果:以下の通り、変換後の配列yは各列とも最小値は0, 最大値は1となった。
[[0. 0. ]
[0.5 0.5]
[1. 1. ]]
[0. 0.]
[1. 1.]
ここで、
mmscaler.fit(x) # xの平均と分散を計算
y = mmscaler.transform(x) # xを変換
としたのは、以下のようにしても同じ結果となる。
y = mmscaler.fit_transform(x) # xを変換した結果が返る
各変換の比較 #
2次元のデータを対象として、スケール変換の効果を散布図で確認する。 各変換のパラメータはデフォルトとした。
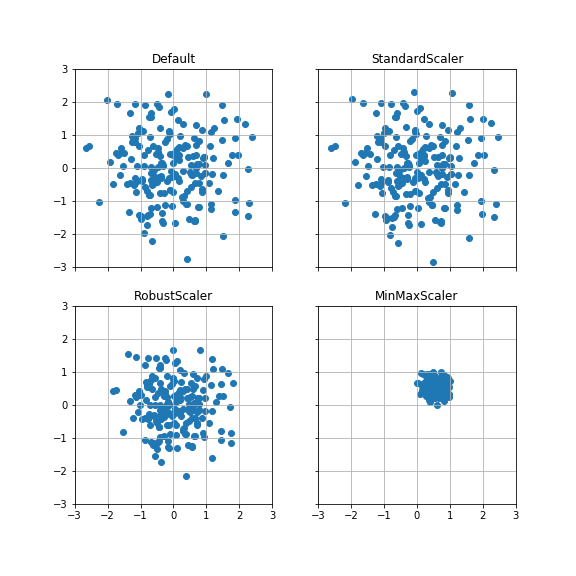
ケース1:平均(0, 0), 分散1 #
平均が原点、分散が各方向に1の正規分布データ200点を変換する。
StandardScalerは、変換前とほとんど変わらない。
RobustScalerは、StandardScalerよりも分散が小さくなっている。
また、MinMaxScalerは縦方向・横方向ともに0~1の範囲に収まっている。
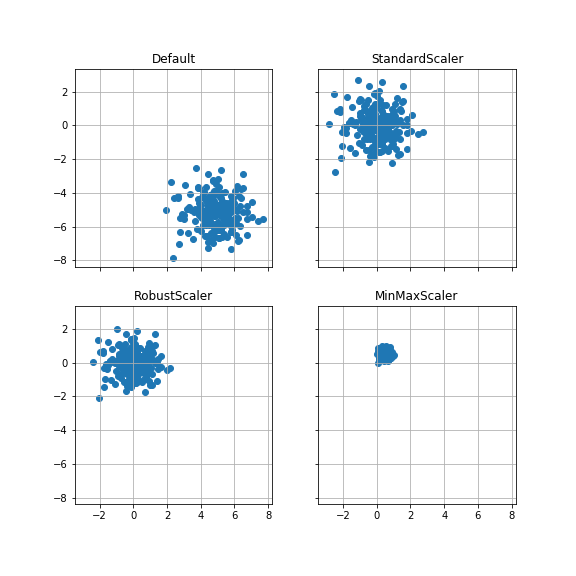
ケース2:平均(5, -5), 分散1 #
平均が(5, -5), 分散が各方向に1の正規分布データ200点を変換する。
StandardScalerは、データの分布形状をほぼ保ったまま変換できている。
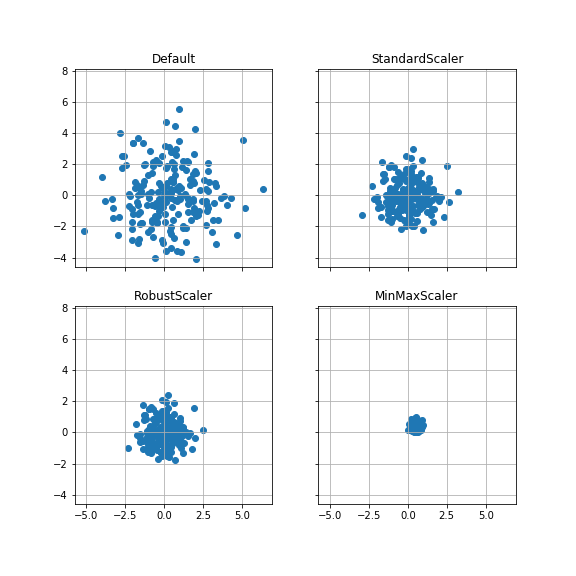
ケース3:平均(0, 0), 分散4 #
平均が(0, 0), 分散が各方向に4とした正規分布データ200点を変換する。
分散が4(標準偏差が2)なので、各方向とも-2~2の範囲に約68.27%, -4~4の範囲に約95.45%のデータがある。
StandardScalerでは分散が約半分となり、-1~1の範囲に約68.27%, -2~2の範囲に約95.45%のデータがあることになる。
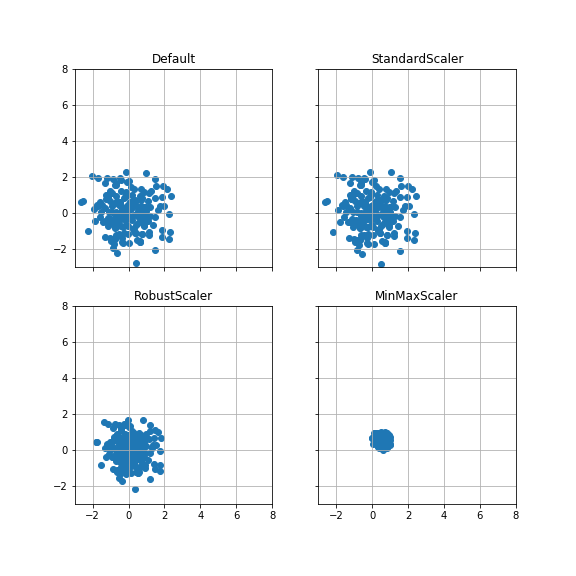
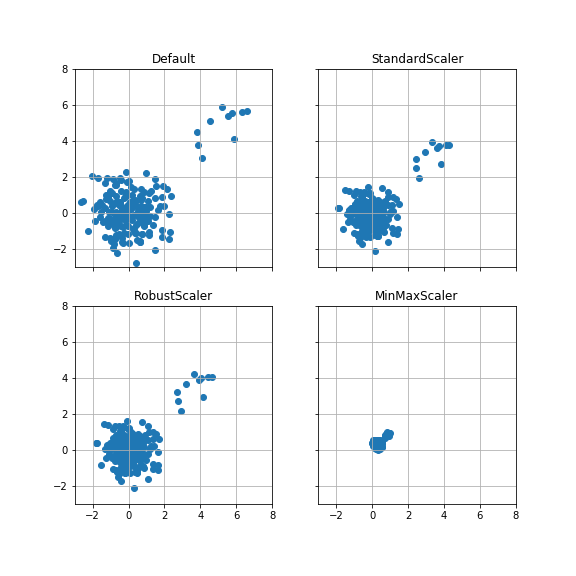
ケース4:外れ値を加えた場合 #
ケース1のデータに、平均(5, 5), 分散1のデータを10点追加する。
ケース1の散布図をスケールを揃えて比較する。
RobustScalerでは、外れ値を除く分布の形状が、外れ値を加える前と後で変化していない。
一方、StandardScaler, MinMaxScalerでは、分布の形状が外れ値によって変化している。
ケース1(外れ値なし)
ケース1~4のソースコードは以下の通り。
def plotScaler(x, xlim=(None, None), ylim=(None, None)):
sscaler = preprocessing.StandardScaler()
rscaler = preprocessing.RobustScaler()
mmscaler = preprocessing.MinMaxScaler()
xs = sscaler.fit_transform(x)
xr = rscaler.fit_transform(x)
xm = mmscaler.fit_transform(x)
fig, ax = plt.subplots(nrows=2, ncols=2, sharex=True, sharey=True, figsize=(8,8))
ax[0,0].scatter(x[:,0], x[:,1])
ax[0,0].set_title("Default")
ax[0,1].scatter(xs[:,0], xs[:,1])
ax[0,1].set_title("StandardScaler")
ax[1,0].scatter(xr[:,0], xr[:,1])
ax[1,0].set_title("RobustScaler")
ax[1,1].scatter(xm[:,0], xm[:,1])
ax[1,1].set_title("MinMaxScaler")
for i in range(ax.shape[0]):
for j in range(ax.shape[1]):
ax[i,j].axis("square")
ax[i,j].grid()
ax[1,1].set_xlim(xlim)
ax[1,1].set_ylim(ylim)
plt.show()
np.random.seed(0)
# ケース1: 平均(0,0), 分散1
x1 = np.random.randn(200, 2)
plotScaler(x1, xlim=(-3, 3), ylim=(-3, 3))
# ケース2: 平均(5,-5), 分散1
x2_0 = np.random.randn(200, 1)+5
x2_1 = np.random.randn(200, 1)-5
x2 = np.hstack([x2_0, x2_1])
plotScaler(x2)
# ケース3: 平均(0,0), 分散4
x3 = np.random.randn(200, 2)*2
plotScaler(x3)
# ケース4: ケース1に(5, 5), 分散1の外れ値を10点追加
x4_0 = np.random.randn(10, 2)+5
x4 = np.vstack([x1, x4_0])
plotScaler(x1, xlim=(-3, 8), ylim=(-3, 8))
plotScaler(x4, xlim=(-3, 8), ylim=(-3, 8))
参考 #
-
sklearn.preprocessing.StandardScaler — scikit-learn 0.24.0 documentation
-
sklearn.preprocessing.RobustScaler — scikit-learn 0.24.0 documentation
-
sklearn.preprocessing.MinMaxScaler — scikit-learn 0.24.0 documentation
